生体情報システム科学専攻 Major of Biological Information Systems
Protein structural biology
 構造生物学 Structural Biology
構造生物学 Structural Biology水口 峰之 Mineyuki Mizuguchi
- TEL : 076-434-7595
- URL : http://www.pha.u-toyama.ac.jp/phypha2/index-j.html
- Keywords : Protein, Three-dimensional structure, Amyloid disease
研究の背景と目的 Background and Purpose of Study
タンパク質は細胞における主要な構成分子であり、また、生体内で起こる全ての過程において重要な働きをしている。タンパク質の機能は立体構造と密接な関係があると考えられており、そのため、タンパク質の立体構造を実験的に決定することは重要なことであると広く認められている。我々は疾患に関連する様々なタンパク質の立体構造について研究している。 いくつかのタンパク質は、ある一定の条件下では正常な立体構造をとることに失敗し、大きな不溶性凝集体へと会合する。アミロイド線維は、高度に組織化されたクロスβ構造を有する線維状の不溶性凝集体であり、アルツハイマー病、プリオン病、トランスサイレチン関連アミロイドーシスといったアミロイド病を引き起こすと考えられている。我々は、タンパク質の立体構造変化とアミロイド線維形成の関係についても研究している。
Proteins are the major molecular components of cells and are involved in every biological process taking place in living systems. Since it is generally assumed that the function of a protein is closely linked to its three-dimensional structure, experimental determination of the protein structures is a matter of high importance. We have been studying the three-dimensional structures of disease-related proteins (Fig. 1). Under some circumstances, several proteins fail to achieve their functional structures and assemble into insoluble aggregate structures. These insoluble protein aggregates include amyloid fibrils, which are fibrous protein aggregates having highly organized cross-β structure (Fig. 2). Amyloid fibrils are known to cause human amyloid disorders, including Alzheimer's disease, prion diseases and transthyretin-related amyloidosis. We also have been studying the relationship between the structural change of proteins and amyloid fibril formation.
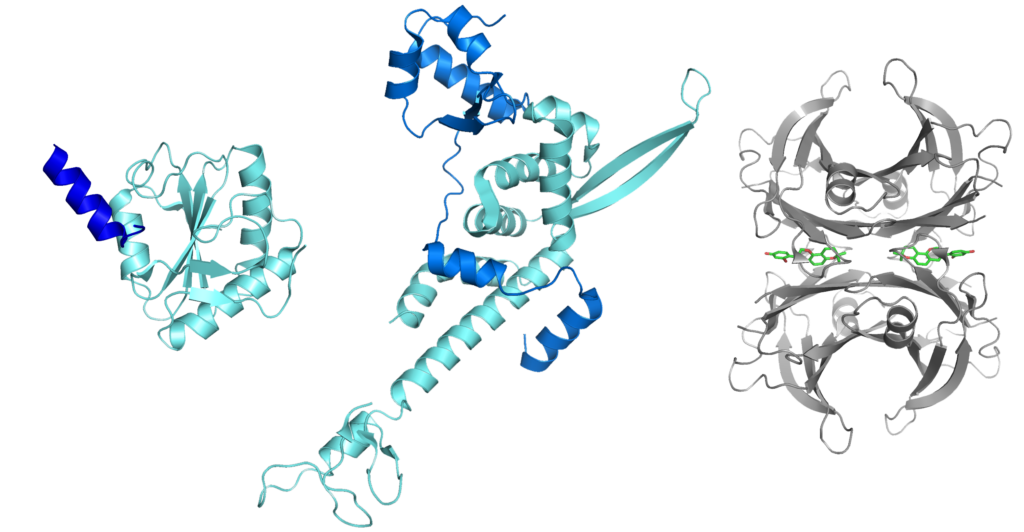
Figure 1. Protein structures determined by X-ray crystallography. (a) PQBP1 bound to spliceosomal U5-15kD (PDB ID: 4CDO) (b) TFIIE complex (PDB ID: 5GPY) (c) V30M mutated transthyretin in complex with glabridin (PDB ID: 4N87).
本研究の領域横断性
タンパク質の立体構造は、タンパク質の機能を原子レベルで理解するために必須であり、医療や産業にとって有用なタンパク質を人工的に創出するタンパク質工学においても重要である。また、タンパク質の立体構造変化とアミロイド線維の関係について理解することは、アミロイド病と総称される病気の治療薬の開発にもつながる。
研究内容
Ⅰ.タンパク質の立体構造解析
我々は、タンパク質の立体構造と機能の関係を理解するために、様々なタンパク質の立体構造について研究している。立体構造解析を行うためには、まず高純度の目的タンパク質をミリグラムのオーダーで大量に得る必要がある。通常我々は大腸菌を使った発現系を構築し、高純度のタンパク質を得ている。立体構造解析は主にX線結晶構造解析と核磁気共鳴(NMR)法を用いて行っている。図1には、近年我々が決定したタンパク質の立体構造を示した。NMR法は、目的タンパク質を結晶化できない場合や、特定の立体構造をとらない天然変性タンパク質について調べる場合に特に有効である。
Ⅱ. タンパク質の立体構造変化とアミロイド線維
生合成されたポリペプチド鎖は、正常な天然構造へと素早く折りたたまれる(フォールディングする)ことでタンパク質としての機能を発揮する。ところが、ポリペプチド鎖が正しくフォールディングすることに失敗し凝集してしまう場合がある。凝集体形成と正常なフォールディングは競合過程であるが、凝集体形成は一般に分子拡散の速度によって律速される遅い反応であるので、通常タンパク質は天然構造へと問題なくフォールディングできる。このことは、タンパク質が効率的に機能を獲得するためには、正常な立体構造へと素早くフォールディングすることが重要であることを示唆している。 タンパク質が凝集すると、しばしば不溶性で分解されにくい線維構造になることがある。このようなタンパク質線維はアミロイド線維と呼ばれ、組織に蓄積してアミロイド病と総称される病気の原因となる。アミロイド病には、アルツハイマー病、プリオン病、家族性アミロイドポリニューロパシーなどが含まれ、それぞれにおいてアミロイド線維を構成するタンパク質は異なっている。
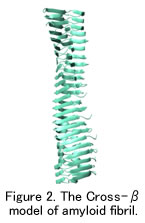 アミロイド病の原因となるタンパク質には、アミノ酸配列や三次元立体構造に相同性がない。しかしながら、これらの原因タンパク質はアミロイド線維になるとクロスβ構造と呼ばれるよく似た形態になる。このモデルでは、アミロイド線維を構成するポリペプチド鎖は線維軸と垂直方向にβストランドとなり、かつ線維軸方向にβシートとなって配置している (Fig. 2)。一般にアミロイド線維は直径約10 nmで枝分かれがなく長さは数μメートルにまで達する。また、プロトフィラメントと呼ばれる2本以上のより細い線維からできており、成熟したアミロイド線維の中で絡みあっている。
アミロイド線維が形成される過程での共通点は、変性ポリペプチドまたは部分的に変性した中間体構造が会合してアミロイド線維になることである。また、多くの場合アミロイド線維形成は核依存的重合モデルで説明できる。このモデルでは、線維構造の鋳型となる種(シード)ができ、そのシードの末端にタンパク質が結合して線維が伸張していく。この過程において律速となるのはシードができる反応であり、いったんシードが形成されると比較的速やかに線維が伸張する。
アミロイド病の原因となるタンパク質には、アミノ酸配列や三次元立体構造に相同性がない。しかしながら、これらの原因タンパク質はアミロイド線維になるとクロスβ構造と呼ばれるよく似た形態になる。このモデルでは、アミロイド線維を構成するポリペプチド鎖は線維軸と垂直方向にβストランドとなり、かつ線維軸方向にβシートとなって配置している (Fig. 2)。一般にアミロイド線維は直径約10 nmで枝分かれがなく長さは数μメートルにまで達する。また、プロトフィラメントと呼ばれる2本以上のより細い線維からできており、成熟したアミロイド線維の中で絡みあっている。
アミロイド線維が形成される過程での共通点は、変性ポリペプチドまたは部分的に変性した中間体構造が会合してアミロイド線維になることである。また、多くの場合アミロイド線維形成は核依存的重合モデルで説明できる。このモデルでは、線維構造の鋳型となる種(シード)ができ、そのシードの末端にタンパク質が結合して線維が伸張していく。この過程において律速となるのはシードができる反応であり、いったんシードが形成されると比較的速やかに線維が伸張する。
参考文献
- Mizuguchi M, Obita T, Serita T, Kojima R, Nabeshima Y, Okazawa H. Mutations in the PQBP1 gene prevent its interaction with the spliceosomal protein U5-15 kD. Nature Commun. 2014, 5, 3822.
- Yokoyama T, Kosaka Y, Mizuguchi M. Crystal structures of human transthyretin complexed with glabridin. J. Med. Chem. 2014, 57, 1090-1096.
- Yokoyama T, Kosaka Y, Mizuguchi M. Inhibitory activities of propolis and its promising component, caffeic acid phenethyl ester, against amyloidogenesis of human transthyretin. J. Med. Chem. 2014, 57, 8928-8935.
- Miwa K, Kojima R, Obita T, Ohkuma Y, Tamura Y, Mizuguchi M. Crystal structure of human general transcription factor TFIIE at atomic resolution. J. Mol. Biol. 2016, 428, 4258-4266.
- Mizuguchi M, Obita T, Kajiyama A, Kozakai Y, Nakai T, Nabeshima Y, Okazawa H. Allosteric modulation of the binding affinity between PQBP1 and the spliceosomal protein U5-15kD. FEBS Lett. 2016;590:2221-2231.

