生体情報システム科学専攻 Major of Biological Information Systems
Regulation of protein degradation
 タンパク質科学 Protein science
タンパク質科学 Protein science伊野部 智由 Tomonao Inobe
- TEL : 076-445-6551
- URL : http://inobelab.wordpress.com
- Keywords : Protein, Protein degradation, Proteasome, Biophysics
研究の背景と目的 Background and Purpose of Study
生命活動の主役を担うタンパク質の量は、合成のみならず、時と場合に応じた分解によっても制御されている。そのためタンパク質の分解異常は、ガンや神経変性疾患など多くの疾患の発症をもたらす。ユビキチン−プロテアソーム系(UPS)は細胞内の重要なタンパク質分解機構であり、不要とされるタンパク質にポリユビキチン鎖を付加し、これを目印としてプロテアソームでの選択的な分解を行うと考えられてきた。我々は、この分解機構による効率的分解にはポリユビキチン鎖の付加だけでは不十分で、分解の標的となるタンパク質自身にフラフラとしたUnstructured領域が必要であることを明らかにした。これらの成果はポリユビキチン鎖ではなく、Unstructured領域がタンパク質の最終的な運命を決めていることを示している。我々は、Unstructured領域を介したプロテアソームによるタンパク質分解の分子メカニズムを解き明かした上で、Unstructured領域に着目した分解の人工制御方法の開発を目指している。
Cellular protein concentrations are controlled through their degradation as well as synthesis. Thus, abnormal protein degradation leads to the onset of various diseases, such as cancer and neurodegenerative diseases. In the eukaryotic cells, most of this degradation is by the ubiquitin proteasome system (UPS). It had been believed that proteins to be degraded by the proteasome must be polyubiquitinated. However, ubiquitination by itself does not always lead to rapid degradation and efficient degradation by the proteasome requires the presence of an unstructured region in the substrate. We determined that nature of unstructured region which induce rapid degradation and propose that the nature of unstructured region determines protein’s fate. Our goal is to elucidate the mechanism of the proteasomal protein degradation and to construct artificial regulation system of the proteasomal degradation by focusing on unstructured degrons.
本研究の領域横断性
生命活動の主役を担うタンパク質の量は、タンパク質合成だけでなく、タンパク質分解によっても調整されている。そのため、我々の行うプロテアソームによる細胞内タンパク質分解の制御に関する研究や、人工分解制御技術の開発は、基礎研究だけでなく、タンパク質量の異常が原因となる病気の理解や治療にもつながる。
研究内容
1.Unstructured領域を介したプロテアソームによるタンパク質分解の仕組み
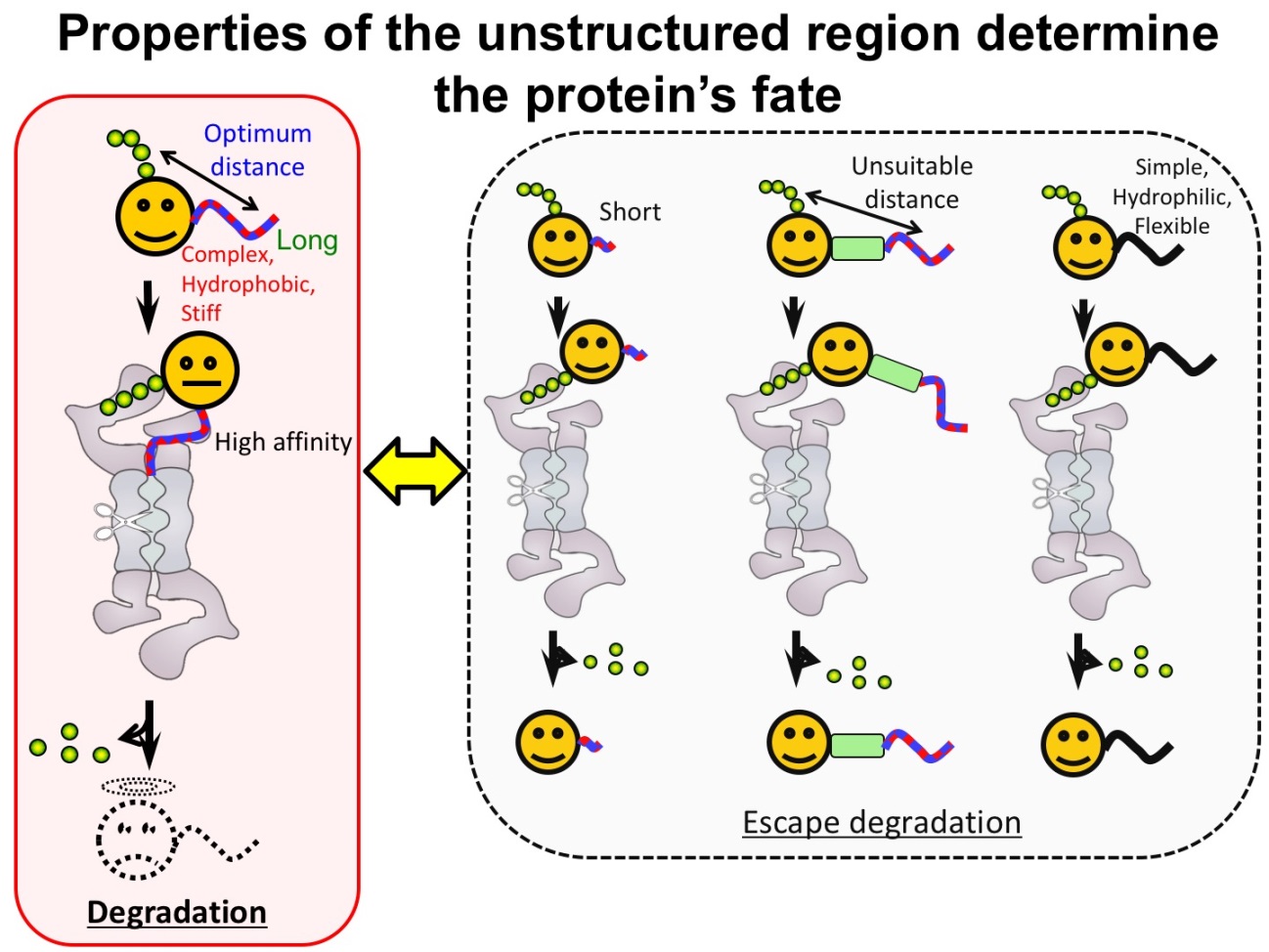
我々は、UPSによるタンパク質分解にはポリユビキチン鎖をシグナルとするプロテアソームへの運び込みだけでは不十分で、標的蛋白質自身にアンフォールド・分解の起点となるUnstructured領域が必要であると提唱した(図1)(Inobe & Matouschek (2014) Curr. Opin. Struct. Biol.)。我々は、以下の研究でこの仮説を証明し、効率的な分解を引き起こすUnstructured領域の特徴を詳細に調べ上げた。 ●効率的な分解を誘導するUnstructured領域の配置や長さ Unstructured領域がある程度以上の長さを持ち、ユビキチンタグから適度な距離にあるときにのみ、効率的な分解が起こる。ユビキチンタグから近すぎても遠すぎても、分解を引き起こすことが出来ない。この配置依存性はポリユビキチン鎖とユビキチン様ドメインの間で異なる。Unstructured領域の位置や配置は、プロテアソームが分解する蛋白質を選ぶにあたり、重要な役割を担っていると考えられる(図1)(論文1)。 ● 効率的な分解を誘導するUnstructured領域のアミノ酸配列 Unstructured領域アミノ酸配列の多くの物理化学的性質は、分解の受けやすさと相関はないが、配列の複雑度は分解の受けやすさと相関がある。つまり複雑なアミノ酸配列から構成されるUnstructured領域をもつ蛋白質だけが、プロテアソームによって分解される(図1)。酵母の生育に必須なE2酵素Cdc34など細胞内の多くの蛋白質も、このルールに従い分解のされやすさが決まっている(論文2)。 ●複合体タンパク質のUnstructured領域を持ったサブユニット特異的な分解 ポリユビキチンタグとUnstructured領域がタンパク質複合体中の2つの別のポリペプチド鎖に分離された場合、プロテアソームはUnstructured領域を持ったサプユニットだけ特異的に分解し、ポリユビキチンタグをもつサブユニットは分解を免れることができる。(図2C参照)(Prakash, Inobe et al.(2009) Nature Chem. Biol.)。 以上の成果は、Unstructured領域がタンパク質の最終的な運命を決めていることを示している。
2.Unstructured領域をターゲットにした分解の制御
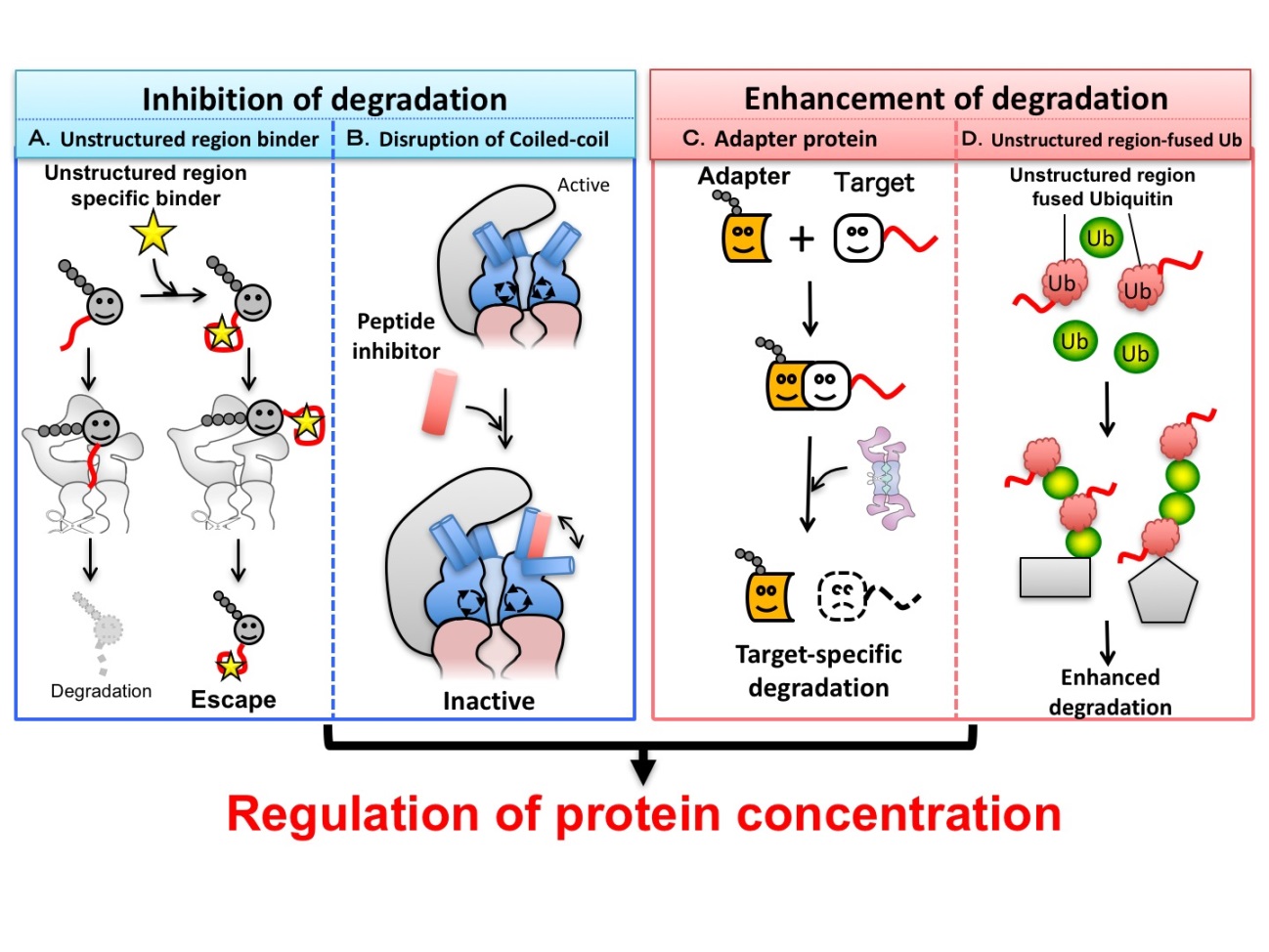
我々は分解の最終決定因子であるUnstructured領域のプロテアソームへのアクセスを加速させたり、阻害したりすることによって、標的タンパク質の特異的分解制御(分解誘導と分解抑制)が可能になる技術の開発を行い、以下のような成果をあげている(図2)。 ● Unstructured領域への競合的な分子結合による分解抑制方法 標的タンパク質のUnstructured領域の性質をリガンド結合や修飾などにより変化させれば、標的タンパク質のプロテアソームによる分解を抑制できることを示した(図2A)(論文3)。この発見を踏まえ、Unstructured領域結合抗体による、より汎用的な標的タンパク質選択的な分解阻害方法の開発を行っている(未発表)。 ● Unstructured領域認識Coiled-coil突起を狙った分解抑制方法 プロテアソームATPaseリングから飛び出たCoiled-coil突起構造は、ATPaseリングの構造を保つためだけでなく、標的タンパク質のUnstructured領域認識に関与していることを明らかにした(論文4)。そしてこのCoiled-coil領域をミミックするペプチドは、本来のCoiled-coil構造を乱し、プロテアソームを阻害すると報告した(図2B)(Inobe & Genmei (2015) Biochem. Biophys. Res. Commun.)。 ● プロテアソームへタンパク質を運び込み分解を誘導するアダプタータンパク質 上記のサブユニット特異的な分解の研究成果を踏まえ、標的タンパク質の分解を誘導するアダプタータンパク質を開発した(図2C)。標的タンパク質を特異的に認識する小型抗体と、プロテアソーム結合標識を融合したアダプタータンパク質は、標的タンパク質をプロテアソームに運び込み、その分解を誘導することを示した(未発表)。 ● Unstructured領域を融合したユビキチンによる分解誘導 変異導入によりUnstructured領域を露出しやすい不安定化ユビキチンは、プロテアソームにより分解されやすい(論文5)。これを踏まえ開発した分解誘導性Unstructured領域融合ユビキチンは、標的タンパク質をポリユビキチン化し、その迅速な分解を誘導することを示した(図2D)(投稿中)。 以上で開発した分解制御方法を応用し、特定タンパク質の分解異常が原因となる病気(癌や神経変性疾患など)の新規治療法の確立に挑む。
参考文献
- Inobe, T., Fishbain, S., Prakash, S. and Matouschek, A. (2011) Defining the Geometry of the Two-component Proteasome Degron. Nature Chem. Biol. 7, 161-167.
- Fishbain, S., Inobe, T., Israeli, E., Chavali, S., Yu, H., Zokarkar, A., Babu, MM., and Matouschek, A. (2015) The sequence composition of disordered regions affects protein half-life by controlling the initiation step of proteasomal degradation. Nature Struct. Mol. Biol. 22, 214-221.
- Takahashi, K., Matouschek, A. and Inobe, T.* (2015) Regulation of proteasomal degradation modulating an unstructured proteasomal initiation region of a substrate. ACS Chem. Biol. 10, 2537–2543.
- Inobe, T.* and Genmei, R. (2015) N-terminal coiled-coil structure of ATPase subunits of 26S proteasome is crucial for proteasome function. PLoS ONE, 10(7): e0134056.
- Inobe, T.* and Nozaki, M. (2016) Proteasomal degradation of damaged polyubiquitin. Biochem. Biophys. Res. Commun. 471, 34-40.

